Talk:Openhackuarium vol. 57 "Bière"
Notes from Hekkcess to prepare next steps
https://hackpad.com/Bience-The-mess-storm-pqiurRcIDUG
Email du lendemain de Kouros
Salut tout le monde,
Merci encore pour le meeting hier, c'était super de pouvoir se poser avec vous et discuter sur la possibilité de la Nébuleuse d'installer un petit espace fermentation&quality control, ainsi que des projets à développer ensemble. On voit qu'il y'a beaucoup à faire, à nous maintenant de mettre en place un roadmap.
1
Le plus simple, le plus rapide à mettre en place, et le plus crucial pour nous actuellement, est de mettre en place un petit programme de propagation + quality control. ==>Time frame: on peut starter asap
Grâce à cela nous allons commencer à accumuler le data nécessaire pour la suite des opérations. Cela prendra un certain temps avant d'avoir accumulé assez de data, sauf si on va de suite à la pêche aux infos ailleurs, type White Labs etc. Ce qui est important c'est donc de définir quels sont les variables que nous rechercherons d'abords et dans quel ordre? Je liste les possibilités et on pourra discuter sur la meilleure marche à suivre.
- Cell Count
==>la théorie sur le nombre de cellules idéales à inoculer est la suivante:
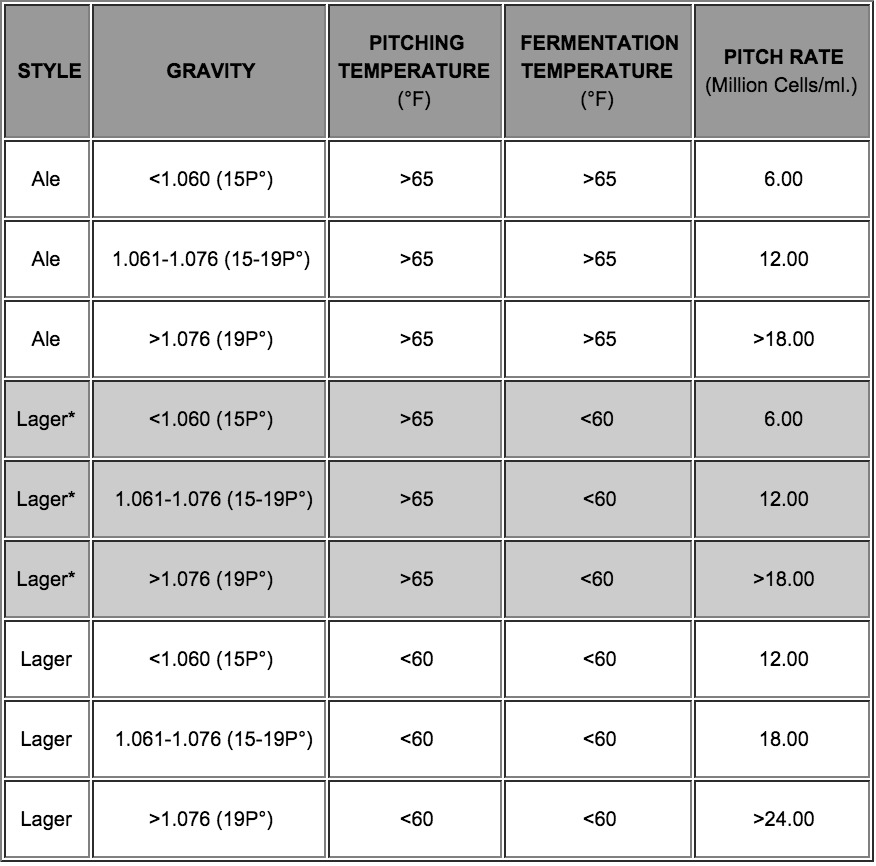
Peut-on approfondir la recherche encore plus loin? Définir pour les températures exacts, pour chaque souche. Y'a-t-il d'autres facteurs qui pourraient influencer le cell count autre que la densité de sucre et la température?
- Mutations ==>selon la théorie, il est possible de réutiliser jusqu'à 15 générations avant que les mutations ne deviennent trop marquées. ==>dans nos conditions de travail actuelles et avec les souches que nous utilisons, à quelle vitesse les mutations se produisent-elles chez nous? Quels sont donc les facteurs augmentant ou réduisant la fréquence de mutation?
- Fermentecibles ==>les chaines de sucres sont plus ou moins fermentecibles (glucose, maltose, fructose, etc). ==>Quelles souches fermentent donc quels sucres à quel taux? Y'aurait-il des facteurs pouvant changer la capacité de fermentation d'un souche?
- Flavour Compounds ==>Nous savons que certaines souches créent certains compounds variants selon les conditions de fermentations. ==>Quantifier ces compounds précisément et les variations selon l'environment
Le mieux serait donc de prendre les sujets de recherche l'un après l'autre et créer un wiki dans lequel on peut amasser les données et les mettre à disposition du publique. En soit il n'est pas possible de définir une date finale sur l'accumulation de ces données vu que les infos sont virtuellement infinies. Il faut donc définir la quantité de data nécessaire pour que les conclusions commencent à devenir concretes.
Nous pouvons aussi imaginer publier des articles sur chacun de ces domaines. Ou alors même créer un "Journal Bience" dans lequel nous informons sur l'avancée du projet et publions des articles.
2
La prochaine étape concerne ensuite le development de projets annexes tels que:
- Génotypage ==>pinpoint les gènes influençant les différents paramètres d'un fermentation ==>nous pouvons aller à la pêche aux gènes (GWAS) ou faire des alignements de séquences (BLAST) ==>cela nous sera aussi utile quand on voudra se lancer dans le développement d'une souche nouvelle ==>L'avantage serait d'entrer en contact avec White Labs et collaborer avec eux sur ce genre d'infos qu'ils ont déjà collecté (contact de Luc) ==>Ces infos peuvent aussi être partagées et recherchées en collaboration avec BeerDecoded
- Breeding ==>un tel projet nécessiterait tout de même plusieurs mois (-->12?) de recherches avant de pouvoir être réalisable. Il nous faudrait ensuite définir quelles sont les caractéristiques recherchées
3
Après plusieurs années de R&D nous pouvons nous atteler au projet final: le software de fermentation. ==>Nous avons encore pas mal de temps pour paufiner ce point là et penser à sa forme. Il faudra avoir accumulé une sacré quantité de data pour que cela devienne crédible.
Donc pour résumer:
Étape 1a - propagation/fermentation/quality control
Time Frame - asap
Étape 1b - Data collection/research topics Time Frame - d'ici la fin de l'année
Étape 2 - Project development Time Frame - deuxième moitié 2016
Étape 3 - Software development Time Frame - pas avant 2017 ?
En terme de matos, dans l'ordre de priorité:
- de la vaisselle de labo, 1L et 10L, des bouchons genre watt qui laissent passer l’oxygène
- plaque chauffante si possible, dans l'idéal un incubateur (assez grand pour les flasque de 10 L.
- Un thermomètre de labo avec une longue tige
- Du milieu de culture (DME) à base de malte
- Des nutriments pour levures genre https://www.wyeastlab.com/hb_productdetail.cfm?ProductID=15
- Du matériel pour compter les cellules : Un spectrophotomètre ou une chambre de comptage (hémocytomètre) et un microscope. (ou la clef USB géniale)
- Un microscope classique 400x
- Du bleu de methylene (Si on a un hémocytomètre)
- Des kits pour asses les infections genre : http://www.merckmillipore.com/CH/fr/product/Kit-de-test-Levures-et-Moisissures,MM_NF-MYSK10025
- S'ils préfèrent faire maison, de l'agar en poudre, un microonde, du glucose de labo
C'est un début pour l'étape 1. Ensuite pour aller plus loin on peut imaginer :
- Des réactifs pour faire des extractions de protéines, d'ADN, voir de mRNA. à nouveau de l'agar en poudre et une machine d'électrophorèse.
- Un thermocycleur PCR
- une taqMan machine pour quantifier l'ARN
Voilà une liste d'idées, à discuter ensemble ce qui peut être sourcé à prix ultra compétitif ;) Il y'a aussi pas mal de petit matos facilement disponible et pas cher (bleu de méthylène. etc?) Il y'a aussi le Fermentor qu'on pourrait mettre en service. Déjà voir ce qu'il peut faire vraiment pour donner une idée de ses capacités.
Quand est-ce qu'on se pose ensemble pour préparer le début ?
J'ai attaché la présa de hier.
Bonne fin de semaine à vous tous et à bientôt!