Difference between revisions of "Bience"
| (41 intermediate revisions by the same user not shown) | |||
| Line 7: | Line 7: | ||
[[File:Bience at work WEsessions.jpg|500px|thumb|none|alt=A| Luc visite la brasserie avec Kouros et Romain pour mieux comprendre le matériel réel utilisé en phase de fermentation.]] | [[File:Bience at work WEsessions.jpg|500px|thumb|none|alt=A| Luc visite la brasserie avec Kouros et Romain pour mieux comprendre le matériel réel utilisé en phase de fermentation.]] | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | == | + | = Techniques utiles à la Bience = |
| − | + | == Vocabulaire et mathématique == | |
| − | + | ===Des chiffres immenses=== | |
| + | Lorsque l'on parle de levure et de tout autre microorganisme, les chiffres deviennent vite bien trop grand pour se les représenter. On utiliser donc la notation scientifique afin de faciliter la communication. Prenons un exemple simple : 1 g de levure sèche contient (selon le fabricant), '''6 000 000 000''' cellules donc '''6 milliards'''. En notation scientifique on note cela '''6*10^9''' qu'on lit comme '''six fois dix puissance neuf''' donc 6 suivit de 9 zéros. Étant donné qu'on monte en générale jusqu'à 10^13, on préfère cette notation. Lorsqu'on parle de chiffre aussi grand, doubler un effectif n'a pas beaucoup de sens. À l'échelle de la levure, passer de 1*10^13 à 2*10^13 ne peut prendre (en théorie et en bonne conditions), que 90 minutes. On se permet donc d'arondir 1.57*10^12 en 1.6*10^12 par exemple. Il est aussi possible de noter cela comme 1.6e+12.<br><br> | ||
| − | + | Une autre idée importante des cultures de levures est le nombre de cellules par millilitre. Ce chiffre represente la concentration d'une culture. Reprenons nos 6*10^9 cellules dans nos 1 g de poudre. Si on les inocule dans 100 ml de milieu, la concentration sera de 6*10^7 Cellules par millilitre (C/ml). Le calcul est simple, on divise le nombre de cellules par le nombre de millilitre. En travaillant avec la notation scientifique, le calcul revient à soustraire 2 (les 2 zéros de 100) à la puissance 9 (9 - 2 = 7). On travaille en général en concentration jusqu'à la dernière culture de 100 L à partir de laquelle on calcul le nombre effectif de levures en multipliant la concentration par 1*10^5 ml. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
== Culture solide == | == Culture solide == | ||
===Principe=== | ===Principe=== | ||
| − | La culture sur plaque est un outil indispensable au bon Bientifique. Ces cultures permettent de répondre à un grand nombre de questions allant du nombre de cellules effectif d'une culture liquide, à la détection et l'identification des infections. Il existe un grand nombre de milieux différents en fonction des besoins. Ici, nous parlerons surtout de ceux que nous utilisons à la Nébuleuse. Principalement, nous utilisons un milieu à base de malt extract (de moût) dans lequel on ajoute du yeast extract maison, de l'agar et une pointe de sulfate de zinc. | + | La culture sur plaque est un outil indispensable au bon Bientifique. Ces cultures permettent de répondre à un grand nombre de questions allant du nombre de cellules effectif d'une culture liquide, à la détection et l'identification des infections. Il existe un grand nombre de milieux différents en fonction des besoins. Ici, nous parlerons surtout de ceux que nous utilisons à la Nébuleuse. Principalement, nous utilisons un milieu à base de malt extract (de moût) dans lequel on ajoute du yeast extract maison, de l'agar et une pointe de sulfate de zinc. Ce milieu très basique permet de faire pousser un peu près tous les organismes trouvés dans une brasserie. Il manque cependant de methode séléctive permettant la démarquation et l'identification des différentes souches. Le milieu LMDA possède ces attout et est décrit dans la partie "Détection d'infections". |
===Milieu basic: Recette=== | ===Milieu basic: Recette=== | ||
| − | ''Il n'est pas nécessaire de travailler en condition stérile à | + | ''Il n'est pas nécessaire de travailler en condition stérile jusqu'à l'étape d'autoclave. La suite doit être stérile.''<br> |
'''À partir de milieu liquide (250 ml)'''<br> | '''À partir de milieu liquide (250 ml)'''<br> | ||
'''1.''' Utiliser un milieu liquide complet d'une gravité d'au moins 8 p° (couper à l'eau déminéralisée si nécessaire pour économiser).<br> | '''1.''' Utiliser un milieu liquide complet d'une gravité d'au moins 8 p° (couper à l'eau déminéralisée si nécessaire pour économiser).<br> | ||
'''2.''' Ajouter 3.75 g d'agar (1.5%) et bien mélanger. Passer eventuellement au microonde une minute pour aider à dissoudre.<br> | '''2.''' Ajouter 3.75 g d'agar (1.5%) et bien mélanger. Passer eventuellement au microonde une minute pour aider à dissoudre.<br> | ||
| − | '''3.''' Autoclaver le milieu puis laisser refroidir à température ambiante avant de stocker au frigo.<br> | + | '''3.''' Autoclaver le milieu puis laisser refroidir à température ambiante avant de stocker au frigo. Il est possible de couler les plaques directement à ce moment. C'est même plus pratique car le milieu est encore liquide en sortant de l'autoclave.<br> |
''Travailler en condition stérile à partir de ce point''<br> | ''Travailler en condition stérile à partir de ce point''<br> | ||
| − | '''4.''' Au moment de couler les plaques, passer au microonde jusqu'à ce que tout le contenu redevienne liquide. Disposer les plaques en cercles autour de la flamme sous la hotte stérile et ajouter | + | '''4.''' Au moment de couler les plaques, (si le milieu était stocké au frigo), passer au microonde jusqu'à ce que tout le contenu redevienne liquide. Disposer les plaques en cercles autour de la flamme sous la hotte stérile et ajouter 10 ml de milieu pour les petites plaques et ~30 ml pour les grandes. Une fois l'agar polymerisé on stocke les plaques fermées et à l'envers au frigo dans un emballage stérile (réutiliser celui des boites). |
== Cultures liquides == | == Cultures liquides == | ||
===Principe=== | ===Principe=== | ||
| − | Les culture liquides sont au coeur du projet Bience. Ces cultures sèrvent principalement à la propagation en masse des levures. Le milieu de culture que l'on utilise est composé de moût (extrait de malte) complété avec du yeast extract et du sulfate de zinc. On commence une culture à partir de levure sèche ou à partir d'une colonie sur culture solide afin d'en obtenir de grandes quantités. Les différentes étapes de 24h se déroulent dans des volumes croissant (1ml, 10 ml, 100 ml, 1 L, 10 L et 100 L). On utilise une gravité d'environ 8 P° jusqu'à 100 ml puis de 12 à 16 P° à partir de 1L. | + | Les culture liquides sont au coeur du projet Bience. Ces cultures sèrvent principalement à la propagation en masse des levures. Le milieu de culture que l'on utilise est composé de moût (extrait de malte) complété avec du yeast extract et du sulfate de zinc. On commence une culture à partir de levure sèche ou à partir d'une colonie sur culture solide afin d'en obtenir de grandes quantités. Les différentes étapes de 24h se déroulent dans des volumes croissant (1ml, 10 ml, 100 ml, 1 L, 10 L et 100 L). On utilise une gravité d'environ 8 P° jusqu'à 100 ml puis de 12 à 16 P° à partir de 1L. Il est possible d'accelérer ces étapes en partant directement de l'étape 10 L avec deux petits sachets de levure solide de 11 g chacun. La première étape consiste donc en 24h dans 10 L, puis 24h dans 100 L avant le pitch dans les cuves de 3000 L. En général, les levures sont capable d'atteindre au moins le même nombre de cellules par millilitre que celui de l'étape d'avant mais dans un volume 10x plus grand. On augmente donc l'effectif de cellules de 10x par étape. Dans les fait, il est possible d'en obtenir plus si l'apport d'air est suffisant. Dans ce protocole accéléré, on commence avec 1.32*10^7 C/ml dans les 10 premiers litres ce qui fait 1.32*10^11 Cellules. (La levure sèche represente environ 6*10^9 C/g). Après 24h sous agitation on peut espérer obtenir au moins 1.32*10^8 C/ml ce qui fait 1.32*10^12 Cellules. On passe ensuite à l'étape 100 L et à la fin on a 1.32*10^8 C/ml donc 1.32*10^13 Cellules ce qui est suffisant pour un pitch correct dans 3000 L. Dans les fait on atteint même ~2*10^13 Cellules (pitch conseillé pour une ale dans 3000 L), mais la différence est négligeable à cette échelle. |
| − | ===Yeast extract : | + | ===Yeast extract : Protocole=== |
| − | Le yeast extract que nous utilisons est obtenu à partir de reste de levures récupéré au moment de la purge des cuves. Le "yeast slurry" est complété avec de l'eau déminéralisée (1:4) et chauffé à 80 C° pendant 2 jours avant d'être autoclavé. Cela a pour effet de lyser les cellules et d'extraire tous les nutriments nécessaires à la croissance des levures. On obtient alors une nette séparation entre les différents débris cellulaires ainsi que les restes de houblon et le yeast extract contenu dans la phase liquide. On récupère le surnageant avec une pipette sérologique au moment de l'utiliser. | + | Le yeast extract que nous utilisons est obtenu à partir de reste de levures récupéré au moment de la purge des cuves. Le "yeast slurry" est complété avec de l'eau déminéralisée (1:4) et chauffé à 80 C° pendant 2 jours avant d'être autoclavé. Cela a pour effet de lyser les cellules et d'extraire tous les nutriments nécessaires à la croissance des levures. On obtient alors une nette séparation entre les différents débris cellulaires ainsi que les restes de houblon et le yeast extract contenu dans la phase liquide. On récupère le surnageant avec une pipette sérologique au moment de l'utiliser. Ce procédé est idéal pour l'étape des 10 L car on utilise qu'un litre de YE à chaque fois. Pour l'étape des 100 L par contre, cela devient vraiment contraignant et nous avons opté pour des nutriment achetés chez Wyeast. |
| + | |||
===Milieu liquide: recette=== | ===Milieu liquide: recette=== | ||
| − | ''1 L''<br> | + | ''1 L (10x plus 10 L)''<br> |
- 100 ml de yeast extract <br> | - 100 ml de yeast extract <br> | ||
- 900 ml de moût (à couper à l'eau si l'on veut une gravité plus basse)<br> | - 900 ml de moût (à couper à l'eau si l'on veut une gravité plus basse)<br> | ||
| − | - Pour le sulfate de zinc, diluer 0.0625 g dans 1 ml d'eau et ajouter | + | - Pour le sulfate de zinc, diluer 0.0625 g dans 1 ml d'eau et ajouter 10 ul de cette solution dans les 1 L de milieu.<br><br> |
Autoclavez le milieu et il sera prêt à l'utilisation une fois refroidi. | Autoclavez le milieu et il sera prêt à l'utilisation une fois refroidi. | ||
| Line 67: | Line 52: | ||
'''3.''' Préparer une plaque de culture solide et noter sur le couvercle les numéros des tubes correspondant à leur dilution. Déposer 5 ul des dilutions voulues en ligne et attendre 5 à 10 minutes que les gouttes sèchent. Déposer la plaque fermée à l'envers dans l'incubateur à 30 C° et attendre que des petites colonies apparaissent. En général, une nuit.<br> | '''3.''' Préparer une plaque de culture solide et noter sur le couvercle les numéros des tubes correspondant à leur dilution. Déposer 5 ul des dilutions voulues en ligne et attendre 5 à 10 minutes que les gouttes sèchent. Déposer la plaque fermée à l'envers dans l'incubateur à 30 C° et attendre que des petites colonies apparaissent. En général, une nuit.<br> | ||
'''4.''' Compter le nombre de colonies à partir de la dilution la plus basse possible. on compte en général les dilutions #4 (10 000x) ou #5 (100 000x). Un comptage d'une cinquantaine à une centaine de colonie donne un résultat plus précis que de compter une dizaine sur la dilution suivante.<br> | '''4.''' Compter le nombre de colonies à partir de la dilution la plus basse possible. on compte en général les dilutions #4 (10 000x) ou #5 (100 000x). Un comptage d'une cinquantaine à une centaine de colonie donne un résultat plus précis que de compter une dizaine sur la dilution suivante.<br> | ||
| − | '''5.''' Calculer selon '''Cells/ml = #colonies*200*dilution'''. Exemple : On compte 76 colonies sur la dilution #4. La culture d'origine contenait donc 76*200*10 000 = 1. | + | '''5.''' Calculer selon '''Cells/ml = #colonies*200*dilution'''. Exemple : On compte 76 colonies sur la dilution #4. La culture d'origine contenait donc 76*200*10 000 = 1.52*10^8 Cells/ml. |
== Comptage au spectrophotomètre == | == Comptage au spectrophotomètre == | ||
| Line 80: | Line 65: | ||
'''4.''' On utilise la même cuvette que le blanc pour mesurer l'échantillon dilué. Il faut mesurer une OD comprise entre 0.3 et 0.8 afin d'être le plus précis possible. Diluer plus ou moins l'échantillon jusqu'à être dans le bon range.<br> | '''4.''' On utilise la même cuvette que le blanc pour mesurer l'échantillon dilué. Il faut mesurer une OD comprise entre 0.3 et 0.8 afin d'être le plus précis possible. Diluer plus ou moins l'échantillon jusqu'à être dans le bon range.<br> | ||
'''5.''' Calculer 3e+6 cellules par OD selon la formule : OD*3e+6*dilution | '''5.''' Calculer 3e+6 cellules par OD selon la formule : OD*3e+6*dilution | ||
| + | |||
| + | == Comptage avec la chambre thoma == | ||
| + | === Principe === | ||
| + | La chambre thoma, ou hemocytomètre, est une lame de microscope spéciale équipée d'une grille de mesure visible à grossissement 10x. Une lamelle concue spécialement pour cette lame permet d'obtenir un puit d'une profondeur connue dans lequel on ajoute ~8 ul d'une culture diluée. La grille permet de compter les cellules présentes et un calcul donne en la concentration cellulaire. | ||
| + | === Protocole === | ||
| + | ''Il n'est pas nécessaire de travailler en condition stérile à par au moment de récupérer l'échantillon dans la culture ou la cuve.''<br> | ||
| + | '''1.''' Récupérer un échantillon à compter et pratiquer une dilution de 100x pour une bière et de 1000x pour une culture. <br> | ||
| + | '''2.''' humidifier les parties latérales de la thoma chamber (avec de l'eau ou de la bave pour les plus téméraires) et poser la lamelle prévue à cet effet en long sur la chambre.<br> | ||
| + | '''3.''' Récupérer ~8 ul de la culture diluée et remplir la chambre en posant la pipette juste au bord de la lamelle. on peut voir le liquide prendre place dans le puit rectangulaire. Il n'est pas nécessaire de vider complétement la pipette, il suffit que le puit soir plein.<br> | ||
| + | '''4.''' Placer la thoma chamber sous le microscope et selon les préférences, compter en grossissement 10x ou 100x. Je préfère personnellement prendre une photo dans l'occulaire en grossissement 10x et compter sur l'écran.<br> | ||
| + | '''5.''' Le comptage se déroule selon la procédure suivante. Séléctionner à l'avance 4 à 5 grands carrés (5*5 petits chacun) et compter le nombre de cellules dans ces grands carrés. Si des cellules sont placée sur une ligne, pas de panique, on compte seulement celles placées sur les lignes des faces haut et droit des carré et on ommet celles des faces bas et gauche.<br> | ||
| + | '''6.''' Calcul : selon la profondeur de la chambre, ce calcul varie. Nous utilisons une chambre "Neubauer improved (depth 0.1mm, square width: 0.05mm)". Le calcul est ''(#CellulesComptée*4e+6)/(#grandsCarrésComptés*25)'' on multiplie ensuite par la dilution (100x ou 1000x) et on obtient le nombre de C/ml de la culture de base. | ||
== Test de fermentation forcée == | == Test de fermentation forcée == | ||
| Line 86: | Line 83: | ||
Pour comprendre ce test, il faut savoir que les levures sont capable de pratiquer la fermentation en condition aérobie (en présence d'oxygène) et anaérobique (sans oxygène). La fermentation aérobie est plus efficace et plus rapide. C'est donc ce système que l'on veut leur faire utiliser. Il suffit de récupérer une petite quantité de moût après le pitch et de le placer dans un erlenmeyer sterile sur agitateur magnétique. La levure, grâce à l'agitation, aura accès à plus d'oxygène que dans la cuve et fermentera le moût plus rapidement. On mesure ensuite la gravité chaque jours jusqu'à ce que la valeur ne varie plus. On sait alors quelle valeur minimal de gravité l'on peut attendre dans la cuve. Une fois atteinte, on peut lancer le cold crash qui provoquera la sédimentation des levures. Attention à ne pas utiliser du matériel contaminé pour ce test. Une autre souche de levure ou de bactérie pourrait être capable de faire descendre la gravité plus bas que la souche présente dans la cuve. | Pour comprendre ce test, il faut savoir que les levures sont capable de pratiquer la fermentation en condition aérobie (en présence d'oxygène) et anaérobique (sans oxygène). La fermentation aérobie est plus efficace et plus rapide. C'est donc ce système que l'on veut leur faire utiliser. Il suffit de récupérer une petite quantité de moût après le pitch et de le placer dans un erlenmeyer sterile sur agitateur magnétique. La levure, grâce à l'agitation, aura accès à plus d'oxygène que dans la cuve et fermentera le moût plus rapidement. On mesure ensuite la gravité chaque jours jusqu'à ce que la valeur ne varie plus. On sait alors quelle valeur minimal de gravité l'on peut attendre dans la cuve. Une fois atteinte, on peut lancer le cold crash qui provoquera la sédimentation des levures. Attention à ne pas utiliser du matériel contaminé pour ce test. Une autre souche de levure ou de bactérie pourrait être capable de faire descendre la gravité plus bas que la souche présente dans la cuve. | ||
| − | == Mesure de gravité == | + | == Mesure de gravité et calcul du yield factor == |
| + | Mesurer la gravité de son moût donne de précieuse informations au brasseur. Cette méthode est aussi bien utile au bientifique qui l'utilise pour mesurer combien les levures ont consommé de sucre dans une culture liquide. Il existe plusieurs méthode de mesure mais à la Nébuleuse, nous utilisons un appareil Anton-paar de mesure de densité. L'important est de savoir quoi faire de cette donnée. Un bon indice de rendement d'un culture est le Yield Factor. On le calcule selon la formule suivante : (C0-Cf)/(G0-Gf) oû C0 est le nombre de milion de cellules au début de la culture et Cf est le nombre de cellules finale en milion aussi, à la fin de la culture. G0 et Gf sont les valeurs de gravité au début et à la fin de la culture. Un yield factor supérieur à 20 indique une croissance aérobique des levures. C'est typiquement le but à atteindre, mais en pratique, sans une bonne aération du milieu avec un agitateur magnetique, il est difficile d'atteindre ce niveau. | ||
| + | |||
== Détection d'infection == | == Détection d'infection == | ||
===Principe=== | ===Principe=== | ||
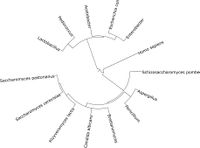
| − | Il est très important d'avoir un moyen de détecter la présence d'autre microorganismes que la levure dans les cultures de levure, dans le moût et dans la bière après fermentation. Certaines bactéries produisent directement des off-flavors d'elles-même comme les lactobacilles alors d'autres induisent les levures présentes à produire des off-flavors. La bière est un environnement hostile pour la plupart des microorganismes (pH bas, éthanol, peu d'oxygène et beaucoup de CO2.) Les principaux microorganismes capable de croître dans la bière sont les suivants : <br><br> | + | Il est très important d'avoir un moyen de détecter la présence d'autre microorganismes que la levure dans les cultures de levure, dans le moût et dans la bière après fermentation. Certaines bactéries produisent directement des off-flavors d'elles-même comme les lactobacilles alors d'autres induisent les levures présentes à produire des off-flavors. La bière est un environnement hostile pour la plupart des microorganismes (pH bas, éthanol, peu d'oxygène et beaucoup de CO2.) Les principaux microorganismes capable de croître dans la bière sont les suivants (liste non exhaustive) : <br><br> |
| + | [[File:pylogenic tree of beer species.jpg|thumb|right|200px|Espèces trouvées dans la bière, qu'elles soient néfastes ou voulues. Séparées ici par distance phylogénétique.]] | ||
'''Gram-Positive Bacteria'''<br> | '''Gram-Positive Bacteria'''<br> | ||
- Lactobacillus spp.<br> | - Lactobacillus spp.<br> | ||
| Line 100: | Line 100: | ||
'''Levures sauvages'''<br> | '''Levures sauvages'''<br> | ||
- Saccharomyces spp.<br> | - Saccharomyces spp.<br> | ||
| − | - Non-Saccharomyces spp. | + | - Non-Saccharomyces spp.<br> |
- Brettanomyces spp.<br> | - Brettanomyces spp.<br> | ||
| − | - | + | - Pichia et Candida spp.<br><br> |
Ces organismes ne présentent pas de danger particulier pour l'homme en bonne santé. Il est cependant important de les éviter dans la bière au risque d'en gâcher le goût. Une infection même légère, sans nécessairement altérer le goût de la bière, peut provoquer une nouvelle fermentation en bouteille et la faire mousser excessivement. Une première indication d'une possible infection se fait au goût et à l'odeur. Une odeur acide indique souvent la présence d'une bactérie. Il est aussi possible de les détécter en mesurant le pH (à condition de déjà connaître le pH final voulu pour une recette donnée). À la Nébuleuse, nous utilisons une petite astuce facilement réalisable en home-brew. Juste avant le pitch, nous gardons 10 à 30 ml de moût prélevé de la cuve. Cela nous permet d'avoir un contrôle sans levure. Nous mesurons la gravité de ce contrôle à la fin de la fermentation afin de s'assurer que toutes les étapes précédant le pitch ont été réalisées de façon stérile. Si une fermentation a eu lieu dans le tube contrôle, il est possible que le moût contenait un microorganisme non voulu et nous lançons les tests de détection.<br><br> | Ces organismes ne présentent pas de danger particulier pour l'homme en bonne santé. Il est cependant important de les éviter dans la bière au risque d'en gâcher le goût. Une infection même légère, sans nécessairement altérer le goût de la bière, peut provoquer une nouvelle fermentation en bouteille et la faire mousser excessivement. Une première indication d'une possible infection se fait au goût et à l'odeur. Une odeur acide indique souvent la présence d'une bactérie. Il est aussi possible de les détécter en mesurant le pH (à condition de déjà connaître le pH final voulu pour une recette donnée). À la Nébuleuse, nous utilisons une petite astuce facilement réalisable en home-brew. Juste avant le pitch, nous gardons 10 à 30 ml de moût prélevé de la cuve. Cela nous permet d'avoir un contrôle sans levure. Nous mesurons la gravité de ce contrôle à la fin de la fermentation afin de s'assurer que toutes les étapes précédant le pitch ont été réalisées de façon stérile. Si une fermentation a eu lieu dans le tube contrôle, il est possible que le moût contenait un microorganisme non voulu et nous lançons les tests de détection.<br><br> | ||
Pour cela, nous utilisons des cultures solide contenant un milieu particulier de détection, largement inspiré du milieu Lee's Multi-Differential Agar (LMDA). Ce milieu contient tous les nutriment nécessaire à la croissance des organismes néfastes à la bière ainsi que du vert de bromocrésol qui joue le role d'indicateur de pH. Le milieu a une couleur bleue de base, pour un pH neutre. Il vire au jaune lorsque le pH baisse, ce qui indique que l'organisme présent produit de l'acide. De plus, la présence du carbonate de calcium insoluble à pH neutre, produit des halos jaunes autour des colonies produisant de l'acide. Nous ajoutons aussi parfois du cycloheximide qui est un puissant antibiotique contre les levure de bière. Cela permet d'isoler les organismes résistants sans détecter la souche de levure voulue. | Pour cela, nous utilisons des cultures solide contenant un milieu particulier de détection, largement inspiré du milieu Lee's Multi-Differential Agar (LMDA). Ce milieu contient tous les nutriment nécessaire à la croissance des organismes néfastes à la bière ainsi que du vert de bromocrésol qui joue le role d'indicateur de pH. Le milieu a une couleur bleue de base, pour un pH neutre. Il vire au jaune lorsque le pH baisse, ce qui indique que l'organisme présent produit de l'acide. De plus, la présence du carbonate de calcium insoluble à pH neutre, produit des halos jaunes autour des colonies produisant de l'acide. Nous ajoutons aussi parfois du cycloheximide qui est un puissant antibiotique contre les levure de bière. Cela permet d'isoler les organismes résistants sans détecter la souche de levure voulue. | ||
| + | |||
===Recette LMDA 250 ml=== | ===Recette LMDA 250 ml=== | ||
- 2.5 g Yeast extract<br> | - 2.5 g Yeast extract<br> | ||
| Line 114: | Line 115: | ||
- 0.025 g de NaCl<br> | - 0.025 g de NaCl<br> | ||
- 0.0055 g de Bromocrésol green (À partir d'une solution concentrée) <br><br> | - 0.0055 g de Bromocrésol green (À partir d'une solution concentrée) <br><br> | ||
| − | Mélanger le tout dans de l'eau déminéralisée et autoclaver la bouteille. On coule ensuite des petites plaques avec 10 ml de milieu. C'est à ce moment (directement dans les plaques) qu'on ajoute ou non le cycloheximide | + | Mélanger le tout dans de l'eau déminéralisée et autoclaver la bouteille. On coule ensuite des petites plaques avec 10 ml de milieu. C'est à ce moment (directement dans les plaques) qu'on ajoute ou non le cycloheximide pour une concentration finale de 0.007 g/l à partir d'une solution préparée précédement. <br> |
| + | |||
===Procédure de test=== | ===Procédure de test=== | ||
'''Méthode'''<br> | '''Méthode'''<br> | ||
| − | Le contenu de la bouteille ou de la cuve suspecte est dilué 10x puis 50 μl sont étalés sur une plaque LMDA contenant du cycloheximide et sur une plaque LMDA sans cycloheximide. Dans le cas d'un test sur une cuve, le contenu du tube contrôle est aussi testé de la même façon. Attendre ensuite 2 jours et analyser les résultats.<br> | + | Le contenu de la bouteille ou de la cuve suspecte est dilué (peut varier selon l'échantillon et peut être adapté selon les résultats) 10x et 1000x puis 50 μl de 10x sont étalés sur une plaque LMDA contenant du cycloheximide et 50 ul de 1000x sur une plaque LMDA sans cycloheximide. Dans le cas d'un test sur une cuve, le contenu du tube contrôle est aussi testé de la même façon. Attendre ensuite 2 jours et analyser les résultats.<br><br> |
'''Interprétation'''<br> | '''Interprétation'''<br> | ||
| − | ''Pas d'infection :'' La plaque sans cycloheximide montre une quantité de colonies attendues et uniformes. Rien ne pousse sur la plaque avec cycloheximide.<br> | + | ''Pas d'infection :'' La plaque sans cycloheximide montre une quantité de colonies attendues et uniformes. Le milieu garde reste bien bleu. Rien ne pousse sur la plaque avec cycloheximide.<br> |
| − | ''Infection bactérienne certaine :'' La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses. La plaque avec cycloheximide montre des colonies.<br> | + | ''Infection bactérienne certaine :'' La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses dont certaines sont entourée d'un halo jaune. La plaque avec cycloheximide montre des colonies dont certaines peuvent montrer un halo jaune ou pas.<br> |
| − | ''Possible infection d'une autre souche de levure :'' La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses. Rien ne pousse sur la plaque avec cycloheximide.<br> | + | ''Possible infection d'une autre souche de levure :'' La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses et/ou un halo jaune apparait autour de certaines colonies. Rien ne pousse sur la plaque avec cycloheximide.<br> |
| − | Dans ce dernier cas de figure une observation au microscope ainsi que de nouvelles plaques en isolant chaque colonie | + | Dans ce dernier cas de figure une observation au microscope ainsi que de nouvelles plaques en isolant chaque colonie est nécessaires pour confirmer une infection. On sait cependant qu'il ne s'agit pas de bactérie car rien n'a poussé avec cycloheximide. Il s'agit donc possiblement d'une autre souche de levure issue d'une cross-contamination ou eventuellement d'une levure sauvage sensible au cycloheximide. |
| − | = | + | = Première expérience : Survie de la S-23 après rehydratation = |
| + | ==Objectif== | ||
| + | Parfois, la levure ne fermente pas bien ou pas assez une fois le moût innoculé. Une des étapes qui n'était pas toujours systèmatique était la réhydratation. Le temps de réhydratation n'était pas toujours le même. Avec cette expérience, nous voulons savoir si le temps de réhydratation a des effets sur la survie. | ||
| + | ==Méthode== | ||
| + | Réhydratation de la S-23 dans 10x le poids en eau. Ici 0.5 g dans 50 ml. Un falcon gardé sous agitation et l'autre sans. Les points sont mesuré à différent temps en récupérant 100 ul de chaque falcon qui sont ensuite dilués 10 000x et inoculé (2x 5 ul) sur des plaques d'agar. les colonies sont compté après 48h d'incubation à 30 C°. Le nombre de cellules vivante est calculé selon la formule : (nombre colonie x 200 x 10 000) | ||
| + | |||
| + | ==Résultat== | ||
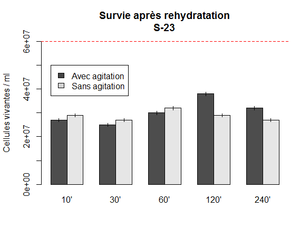
| + | [[File:S-23_survie2.png|300px|thumb|right|'''Fig. 1.''' Survie de la S-23 après réhydratation. Nombre de cellules vivantes par ml en fonction de différents temps de réhydratation. La ligne rouge représente la quantité théorique en fonction du poids de levure sèche utilisée.]] | ||
| + | On observe un pic à 2h sous agitation. Ce que cette expérience indique surtout, est que le temps de rehydratation n'a pas une très grande influence sur la survie. Il est donc possible de garder les levures dans le yeast tank au moins pendant 4 heures sans effet néfaste. | ||
| − | == | + | = Prototype pour mesurer un volume de gas = |
| − | + | ==Objectif== | |
| − | === | + | Pour pouvoir suivre la production de CO2 durant la fermentation, un instrument de mesure du volume de gas est nécessaire. L'objectif de ce prototype est de permettre de tester différentes configurations low-cost et low-tech et d'en comparer la précision. |
| − | + | ==Méthode== | |
| + | Construire un instrument de mesure du volume de gas produit par une culture de levure en fermentation pendant un temps donné. | ||
| + | ==Résultat== | ||
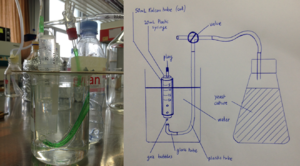
| + | Le prototype 1 est décrit dans le schéma ci-dessous, ainsi que représenté par une photographie | ||
| − | = | + | [[File:Bubbler_prototype1.png|thumb|none|alt=A| This is what the whole setup looks like.]] |
| − | |||
| − | |||
| − | + | [[Category:Work In Progress]] | |
| − | + | [[Category:Projects]] | |
Latest revision as of 13:25, 24 March 2016
Bience (Bière et Science !) est un projet de R&D en microbiologie
Ce projet très sérieux a été initié par nos voisins, les brasseurs de La Nébuleuse, qui veulent mieux comprendre comment fonctionnent les levures qu’ils utilisent pour fabriquer leurs produits. Avec l’aide des biologistes de l’association, ils vont apprendre à dompter ces petites bêtes à Hackuarium.
Le projet a été présenté lors de l' Openhackuarium vol. 57 "Bière" (pas mal de détails sur la page discussion : Talk:Openhackuarium_vol._57_"Bière")
Premiers WE de travail
Techniques utiles à la Bience
Vocabulaire et mathématique
Des chiffres immenses
Lorsque l'on parle de levure et de tout autre microorganisme, les chiffres deviennent vite bien trop grand pour se les représenter. On utiliser donc la notation scientifique afin de faciliter la communication. Prenons un exemple simple : 1 g de levure sèche contient (selon le fabricant), 6 000 000 000 cellules donc 6 milliards. En notation scientifique on note cela 6*10^9 qu'on lit comme six fois dix puissance neuf donc 6 suivit de 9 zéros. Étant donné qu'on monte en générale jusqu'à 10^13, on préfère cette notation. Lorsqu'on parle de chiffre aussi grand, doubler un effectif n'a pas beaucoup de sens. À l'échelle de la levure, passer de 1*10^13 à 2*10^13 ne peut prendre (en théorie et en bonne conditions), que 90 minutes. On se permet donc d'arondir 1.57*10^12 en 1.6*10^12 par exemple. Il est aussi possible de noter cela comme 1.6e+12.
Une autre idée importante des cultures de levures est le nombre de cellules par millilitre. Ce chiffre represente la concentration d'une culture. Reprenons nos 6*10^9 cellules dans nos 1 g de poudre. Si on les inocule dans 100 ml de milieu, la concentration sera de 6*10^7 Cellules par millilitre (C/ml). Le calcul est simple, on divise le nombre de cellules par le nombre de millilitre. En travaillant avec la notation scientifique, le calcul revient à soustraire 2 (les 2 zéros de 100) à la puissance 9 (9 - 2 = 7). On travaille en général en concentration jusqu'à la dernière culture de 100 L à partir de laquelle on calcul le nombre effectif de levures en multipliant la concentration par 1*10^5 ml.
Culture solide
Principe
La culture sur plaque est un outil indispensable au bon Bientifique. Ces cultures permettent de répondre à un grand nombre de questions allant du nombre de cellules effectif d'une culture liquide, à la détection et l'identification des infections. Il existe un grand nombre de milieux différents en fonction des besoins. Ici, nous parlerons surtout de ceux que nous utilisons à la Nébuleuse. Principalement, nous utilisons un milieu à base de malt extract (de moût) dans lequel on ajoute du yeast extract maison, de l'agar et une pointe de sulfate de zinc. Ce milieu très basique permet de faire pousser un peu près tous les organismes trouvés dans une brasserie. Il manque cependant de methode séléctive permettant la démarquation et l'identification des différentes souches. Le milieu LMDA possède ces attout et est décrit dans la partie "Détection d'infections".
Milieu basic: Recette
Il n'est pas nécessaire de travailler en condition stérile jusqu'à l'étape d'autoclave. La suite doit être stérile.
À partir de milieu liquide (250 ml)
1. Utiliser un milieu liquide complet d'une gravité d'au moins 8 p° (couper à l'eau déminéralisée si nécessaire pour économiser).
2. Ajouter 3.75 g d'agar (1.5%) et bien mélanger. Passer eventuellement au microonde une minute pour aider à dissoudre.
3. Autoclaver le milieu puis laisser refroidir à température ambiante avant de stocker au frigo. Il est possible de couler les plaques directement à ce moment. C'est même plus pratique car le milieu est encore liquide en sortant de l'autoclave.
Travailler en condition stérile à partir de ce point
4. Au moment de couler les plaques, (si le milieu était stocké au frigo), passer au microonde jusqu'à ce que tout le contenu redevienne liquide. Disposer les plaques en cercles autour de la flamme sous la hotte stérile et ajouter 10 ml de milieu pour les petites plaques et ~30 ml pour les grandes. Une fois l'agar polymerisé on stocke les plaques fermées et à l'envers au frigo dans un emballage stérile (réutiliser celui des boites).
Cultures liquides
Principe
Les culture liquides sont au coeur du projet Bience. Ces cultures sèrvent principalement à la propagation en masse des levures. Le milieu de culture que l'on utilise est composé de moût (extrait de malte) complété avec du yeast extract et du sulfate de zinc. On commence une culture à partir de levure sèche ou à partir d'une colonie sur culture solide afin d'en obtenir de grandes quantités. Les différentes étapes de 24h se déroulent dans des volumes croissant (1ml, 10 ml, 100 ml, 1 L, 10 L et 100 L). On utilise une gravité d'environ 8 P° jusqu'à 100 ml puis de 12 à 16 P° à partir de 1L. Il est possible d'accelérer ces étapes en partant directement de l'étape 10 L avec deux petits sachets de levure solide de 11 g chacun. La première étape consiste donc en 24h dans 10 L, puis 24h dans 100 L avant le pitch dans les cuves de 3000 L. En général, les levures sont capable d'atteindre au moins le même nombre de cellules par millilitre que celui de l'étape d'avant mais dans un volume 10x plus grand. On augmente donc l'effectif de cellules de 10x par étape. Dans les fait, il est possible d'en obtenir plus si l'apport d'air est suffisant. Dans ce protocole accéléré, on commence avec 1.32*10^7 C/ml dans les 10 premiers litres ce qui fait 1.32*10^11 Cellules. (La levure sèche represente environ 6*10^9 C/g). Après 24h sous agitation on peut espérer obtenir au moins 1.32*10^8 C/ml ce qui fait 1.32*10^12 Cellules. On passe ensuite à l'étape 100 L et à la fin on a 1.32*10^8 C/ml donc 1.32*10^13 Cellules ce qui est suffisant pour un pitch correct dans 3000 L. Dans les fait on atteint même ~2*10^13 Cellules (pitch conseillé pour une ale dans 3000 L), mais la différence est négligeable à cette échelle.
Yeast extract : Protocole
Le yeast extract que nous utilisons est obtenu à partir de reste de levures récupéré au moment de la purge des cuves. Le "yeast slurry" est complété avec de l'eau déminéralisée (1:4) et chauffé à 80 C° pendant 2 jours avant d'être autoclavé. Cela a pour effet de lyser les cellules et d'extraire tous les nutriments nécessaires à la croissance des levures. On obtient alors une nette séparation entre les différents débris cellulaires ainsi que les restes de houblon et le yeast extract contenu dans la phase liquide. On récupère le surnageant avec une pipette sérologique au moment de l'utiliser. Ce procédé est idéal pour l'étape des 10 L car on utilise qu'un litre de YE à chaque fois. Pour l'étape des 100 L par contre, cela devient vraiment contraignant et nous avons opté pour des nutriment achetés chez Wyeast.
Milieu liquide: recette
1 L (10x plus 10 L)
- 100 ml de yeast extract
- 900 ml de moût (à couper à l'eau si l'on veut une gravité plus basse)
- Pour le sulfate de zinc, diluer 0.0625 g dans 1 ml d'eau et ajouter 10 ul de cette solution dans les 1 L de milieu.
Autoclavez le milieu et il sera prêt à l'utilisation une fois refroidi.
Comptage sur plaque
Principe
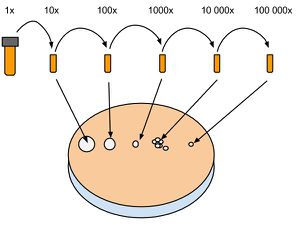
Le comptage sur plaque permet d'avoir une bonne estimation du nombre de cellules dans une culture liquide. Contrairement au comptage au spectrophotomètre, il est nécessaire d'attendre une nuit complète avant d'avoir le résultat. Cette technique donne un résultat plus fiable que le spectrophotomètre. Il est donc préférable de se fier au résultat sur plaque après 24h. La méthode consiste à effectuer une dilution en série d'une culture dont on aimerait connaître le nombre de cellules. Ensuite, 5 ul de culture dilué sont déposé sur une plaque de Culture solide. Le principe est que ces 5 ul contiennent un nombre de cellules suffisamment petit pour que chacune d'elle se divise en une colonie distincte. Les colonies sont ensuite comptées et le nombre de cellule d'origine est calculé selon la formule suivante : Cells/ml = #colonies*200*dilution.
Protocole détaillé
Cette technique nécessite de travailler sous la hotte de ventilation avec du matériel stérile.
1. Préparer 4 à 6 alicots (1.5 ml) selon la concentration estimée, en ligne sur un portoir et les numéroter. Ajouter 900 ml d'H2O stérile dans chaque tube.
2. Ouvrir la culture liquide à mesurer et récupérer 100 ul qu'on ajoute au tube numéro 1 (dilution 10x). Mélanger up and down avec la pipette et récupérer 100 ul à passer dans le tube numéro 2 (dilution 100x) et ainsi de suite.
3. Préparer une plaque de culture solide et noter sur le couvercle les numéros des tubes correspondant à leur dilution. Déposer 5 ul des dilutions voulues en ligne et attendre 5 à 10 minutes que les gouttes sèchent. Déposer la plaque fermée à l'envers dans l'incubateur à 30 C° et attendre que des petites colonies apparaissent. En général, une nuit.
4. Compter le nombre de colonies à partir de la dilution la plus basse possible. on compte en général les dilutions #4 (10 000x) ou #5 (100 000x). Un comptage d'une cinquantaine à une centaine de colonie donne un résultat plus précis que de compter une dizaine sur la dilution suivante.
5. Calculer selon Cells/ml = #colonies*200*dilution. Exemple : On compte 76 colonies sur la dilution #4. La culture d'origine contenait donc 76*200*10 000 = 1.52*10^8 Cells/ml.
Comptage au spectrophotomètre
Principe
Le spectrophotomètre permet de mesurer la densité optique (OD) d'une solution. En réglant la longueur d'onde à 600 nm, il est possible d'avoir une bonne idée de la densité cellulaire d'une culture liquide. Cette mesure n'est pas très précise mais donne une bonne idée de l'ordre de grandeur et permet d'avoir un résultat rapide. On l'utilise en général avant de pitcher afin de déterminer le volume nécessaire. On confirme le résultat avec un comptage sur plaque. Il est nécessaire de diluer 50x à 100x une culture classique (1e+8 C/ml). On utilise donc les première ou deuxième dilutions de la série utilisée pour le comptage sur plaque.
Protocole détaillé
Il n'est pas necessaire de travailler en condition stérile une fois l'échantillon récupéré.
1. Allumer le spectrophotomètre afin de laisser la lampe chauffer. Il faut attendre au moins 15 minutes.
2. Effectuer une dilution jusqu'à obtenir une solution bien translucide (50 à 100x)
3. Prendre une cuvette adaptée au spectro et la remplir d'eau afin de l'utiliser comme "blanc". placer la cuvette dans la machine réglée à 600 nm et appuyer sur le bouton representant une cuvette pour calibrer.
4. On utilise la même cuvette que le blanc pour mesurer l'échantillon dilué. Il faut mesurer une OD comprise entre 0.3 et 0.8 afin d'être le plus précis possible. Diluer plus ou moins l'échantillon jusqu'à être dans le bon range.
5. Calculer 3e+6 cellules par OD selon la formule : OD*3e+6*dilution
Comptage avec la chambre thoma
Principe
La chambre thoma, ou hemocytomètre, est une lame de microscope spéciale équipée d'une grille de mesure visible à grossissement 10x. Une lamelle concue spécialement pour cette lame permet d'obtenir un puit d'une profondeur connue dans lequel on ajoute ~8 ul d'une culture diluée. La grille permet de compter les cellules présentes et un calcul donne en la concentration cellulaire.
Protocole
Il n'est pas nécessaire de travailler en condition stérile à par au moment de récupérer l'échantillon dans la culture ou la cuve.
1. Récupérer un échantillon à compter et pratiquer une dilution de 100x pour une bière et de 1000x pour une culture.
2. humidifier les parties latérales de la thoma chamber (avec de l'eau ou de la bave pour les plus téméraires) et poser la lamelle prévue à cet effet en long sur la chambre.
3. Récupérer ~8 ul de la culture diluée et remplir la chambre en posant la pipette juste au bord de la lamelle. on peut voir le liquide prendre place dans le puit rectangulaire. Il n'est pas nécessaire de vider complétement la pipette, il suffit que le puit soir plein.
4. Placer la thoma chamber sous le microscope et selon les préférences, compter en grossissement 10x ou 100x. Je préfère personnellement prendre une photo dans l'occulaire en grossissement 10x et compter sur l'écran.
5. Le comptage se déroule selon la procédure suivante. Séléctionner à l'avance 4 à 5 grands carrés (5*5 petits chacun) et compter le nombre de cellules dans ces grands carrés. Si des cellules sont placée sur une ligne, pas de panique, on compte seulement celles placées sur les lignes des faces haut et droit des carré et on ommet celles des faces bas et gauche.
6. Calcul : selon la profondeur de la chambre, ce calcul varie. Nous utilisons une chambre "Neubauer improved (depth 0.1mm, square width: 0.05mm)". Le calcul est (#CellulesComptée*4e+6)/(#grandsCarrésComptés*25) on multiplie ensuite par la dilution (100x ou 1000x) et on obtient le nombre de C/ml de la culture de base.
Test de fermentation forcée
Il est nécessaire de travailler en conditions stériles
Le test de fermentation forcée permet d'avoir une idée de l'atténuation à atteindre pour une recette donnée. La valeur de gravité d'un moût peut descendre jusqu'à un certain point en fonction de la souche de levure utilisée. Ce test permet de determiner la valeur de ce point. Il est important de connaître cette valeur afin de ne pas laisser les levures trop longtemps une fois leurs ressources épuisées. C'est à ce moment là, en condition de stress, qu'elles relâchent des métabolites pouvant altérer le goût de la bière. Dans l'autre sens, il est important de ne pas embouteiller alors que cette valeur n'est pas encore atteinte au risque d'avoir une surpression dans les bouteilles.
Pour comprendre ce test, il faut savoir que les levures sont capable de pratiquer la fermentation en condition aérobie (en présence d'oxygène) et anaérobique (sans oxygène). La fermentation aérobie est plus efficace et plus rapide. C'est donc ce système que l'on veut leur faire utiliser. Il suffit de récupérer une petite quantité de moût après le pitch et de le placer dans un erlenmeyer sterile sur agitateur magnétique. La levure, grâce à l'agitation, aura accès à plus d'oxygène que dans la cuve et fermentera le moût plus rapidement. On mesure ensuite la gravité chaque jours jusqu'à ce que la valeur ne varie plus. On sait alors quelle valeur minimal de gravité l'on peut attendre dans la cuve. Une fois atteinte, on peut lancer le cold crash qui provoquera la sédimentation des levures. Attention à ne pas utiliser du matériel contaminé pour ce test. Une autre souche de levure ou de bactérie pourrait être capable de faire descendre la gravité plus bas que la souche présente dans la cuve.
Mesure de gravité et calcul du yield factor
Mesurer la gravité de son moût donne de précieuse informations au brasseur. Cette méthode est aussi bien utile au bientifique qui l'utilise pour mesurer combien les levures ont consommé de sucre dans une culture liquide. Il existe plusieurs méthode de mesure mais à la Nébuleuse, nous utilisons un appareil Anton-paar de mesure de densité. L'important est de savoir quoi faire de cette donnée. Un bon indice de rendement d'un culture est le Yield Factor. On le calcule selon la formule suivante : (C0-Cf)/(G0-Gf) oû C0 est le nombre de milion de cellules au début de la culture et Cf est le nombre de cellules finale en milion aussi, à la fin de la culture. G0 et Gf sont les valeurs de gravité au début et à la fin de la culture. Un yield factor supérieur à 20 indique une croissance aérobique des levures. C'est typiquement le but à atteindre, mais en pratique, sans une bonne aération du milieu avec un agitateur magnetique, il est difficile d'atteindre ce niveau.
Détection d'infection
Principe
Il est très important d'avoir un moyen de détecter la présence d'autre microorganismes que la levure dans les cultures de levure, dans le moût et dans la bière après fermentation. Certaines bactéries produisent directement des off-flavors d'elles-même comme les lactobacilles alors d'autres induisent les levures présentes à produire des off-flavors. La bière est un environnement hostile pour la plupart des microorganismes (pH bas, éthanol, peu d'oxygène et beaucoup de CO2.) Les principaux microorganismes capable de croître dans la bière sont les suivants (liste non exhaustive) :
Gram-Positive Bacteria
- Lactobacillus spp.
- Pediococcus spp.
Gram-Negative Bacteria
- Acetic Acid Bacteria
- Zymomonas spp.
- Pectinatus spp.
- Enterobacteriaceae spp.
Levures sauvages
- Saccharomyces spp.
- Non-Saccharomyces spp.
- Brettanomyces spp.
- Pichia et Candida spp.
Ces organismes ne présentent pas de danger particulier pour l'homme en bonne santé. Il est cependant important de les éviter dans la bière au risque d'en gâcher le goût. Une infection même légère, sans nécessairement altérer le goût de la bière, peut provoquer une nouvelle fermentation en bouteille et la faire mousser excessivement. Une première indication d'une possible infection se fait au goût et à l'odeur. Une odeur acide indique souvent la présence d'une bactérie. Il est aussi possible de les détécter en mesurant le pH (à condition de déjà connaître le pH final voulu pour une recette donnée). À la Nébuleuse, nous utilisons une petite astuce facilement réalisable en home-brew. Juste avant le pitch, nous gardons 10 à 30 ml de moût prélevé de la cuve. Cela nous permet d'avoir un contrôle sans levure. Nous mesurons la gravité de ce contrôle à la fin de la fermentation afin de s'assurer que toutes les étapes précédant le pitch ont été réalisées de façon stérile. Si une fermentation a eu lieu dans le tube contrôle, il est possible que le moût contenait un microorganisme non voulu et nous lançons les tests de détection.
Pour cela, nous utilisons des cultures solide contenant un milieu particulier de détection, largement inspiré du milieu Lee's Multi-Differential Agar (LMDA). Ce milieu contient tous les nutriment nécessaire à la croissance des organismes néfastes à la bière ainsi que du vert de bromocrésol qui joue le role d'indicateur de pH. Le milieu a une couleur bleue de base, pour un pH neutre. Il vire au jaune lorsque le pH baisse, ce qui indique que l'organisme présent produit de l'acide. De plus, la présence du carbonate de calcium insoluble à pH neutre, produit des halos jaunes autour des colonies produisant de l'acide. Nous ajoutons aussi parfois du cycloheximide qui est un puissant antibiotique contre les levure de bière. Cela permet d'isoler les organismes résistants sans détecter la souche de levure voulue.
Recette LMDA 250 ml
- 2.5 g Yeast extract
- 2.5 g Glucose
- 1.25 g Calcium carbonate (Craie blanche réduite en poudre)
- 3.75 g D'agar
- 0.025 g de NaCl
- 0.0055 g de Bromocrésol green (À partir d'une solution concentrée)
Mélanger le tout dans de l'eau déminéralisée et autoclaver la bouteille. On coule ensuite des petites plaques avec 10 ml de milieu. C'est à ce moment (directement dans les plaques) qu'on ajoute ou non le cycloheximide pour une concentration finale de 0.007 g/l à partir d'une solution préparée précédement.
Procédure de test
Méthode
Le contenu de la bouteille ou de la cuve suspecte est dilué (peut varier selon l'échantillon et peut être adapté selon les résultats) 10x et 1000x puis 50 μl de 10x sont étalés sur une plaque LMDA contenant du cycloheximide et 50 ul de 1000x sur une plaque LMDA sans cycloheximide. Dans le cas d'un test sur une cuve, le contenu du tube contrôle est aussi testé de la même façon. Attendre ensuite 2 jours et analyser les résultats.
Interprétation
Pas d'infection : La plaque sans cycloheximide montre une quantité de colonies attendues et uniformes. Le milieu garde reste bien bleu. Rien ne pousse sur la plaque avec cycloheximide.
Infection bactérienne certaine : La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses dont certaines sont entourée d'un halo jaune. La plaque avec cycloheximide montre des colonies dont certaines peuvent montrer un halo jaune ou pas.
Possible infection d'une autre souche de levure : La plaque sans cycloheximide montre un trop grand nombre de colonies de formes diverses et/ou un halo jaune apparait autour de certaines colonies. Rien ne pousse sur la plaque avec cycloheximide.
Dans ce dernier cas de figure une observation au microscope ainsi que de nouvelles plaques en isolant chaque colonie est nécessaires pour confirmer une infection. On sait cependant qu'il ne s'agit pas de bactérie car rien n'a poussé avec cycloheximide. Il s'agit donc possiblement d'une autre souche de levure issue d'une cross-contamination ou eventuellement d'une levure sauvage sensible au cycloheximide.
Première expérience : Survie de la S-23 après rehydratation
Objectif
Parfois, la levure ne fermente pas bien ou pas assez une fois le moût innoculé. Une des étapes qui n'était pas toujours systèmatique était la réhydratation. Le temps de réhydratation n'était pas toujours le même. Avec cette expérience, nous voulons savoir si le temps de réhydratation a des effets sur la survie.
Méthode
Réhydratation de la S-23 dans 10x le poids en eau. Ici 0.5 g dans 50 ml. Un falcon gardé sous agitation et l'autre sans. Les points sont mesuré à différent temps en récupérant 100 ul de chaque falcon qui sont ensuite dilués 10 000x et inoculé (2x 5 ul) sur des plaques d'agar. les colonies sont compté après 48h d'incubation à 30 C°. Le nombre de cellules vivante est calculé selon la formule : (nombre colonie x 200 x 10 000)
Résultat
On observe un pic à 2h sous agitation. Ce que cette expérience indique surtout, est que le temps de rehydratation n'a pas une très grande influence sur la survie. Il est donc possible de garder les levures dans le yeast tank au moins pendant 4 heures sans effet néfaste.
Prototype pour mesurer un volume de gas
Objectif
Pour pouvoir suivre la production de CO2 durant la fermentation, un instrument de mesure du volume de gas est nécessaire. L'objectif de ce prototype est de permettre de tester différentes configurations low-cost et low-tech et d'en comparer la précision.
Méthode
Construire un instrument de mesure du volume de gas produit par une culture de levure en fermentation pendant un temps donné.
Résultat
Le prototype 1 est décrit dans le schéma ci-dessous, ainsi que représenté par une photographie